[J1-基因测序基础知识] 全基因组测序与覆盖深度
1 测序深度概念
Coverage ratio(覆盖比率,亦简称覆盖率,亦称基因组覆盖率),指被测序到的碱基占全基因组大小的比率。
Coverage depth (覆盖深度,亦称测序深度,或者碱基平均测序深度),指每个碱基被测序的平均次数。即测序的数据总量比基因组大小
2 测序深度计算公式
第二代高通量测序的迅速发展的同时,也带来了新的名词,由于这些名词尚无特别明确的定义,因此,对计算公式的定义可对评估及后续的理解带来进一步的帮助。
平均测序深度:测序所得的碱基总数(raw data or clean data)/基因组大小
有效平均测序深度(mapped depth):比对上基因组的数据/去N区后的基因组大小。
3 测序深度与有效测序深度
1. 测序所得的数据(raw data 或clean data)易定量定性,测出来的数据真实反应样品及文库信息。
2. Mapped data不易定量,因为比对受不同的因素影响:
a. 不同的物种比对差异非常大.
b. 同样的物种,不同的样本差异非常大,如部分肿瘤样品、FFPE样品比对率会出现异常情况。
c. 样品的取材部门及环境可能会影响比对结果。样品污染也可能会造成比对率大幅度下降。在没有比对前,我们不可能很清楚样品的具体信息。如唾液样本,其比对率就会下降非常多,其原因就是有杂菌污染
d. 不同的比对软件和参数设置可能导致比对率差异。如相同的数据,BWA与SOAPalign相比,map-rate要多出2%以上。
4 不同平均覆盖深度下的单碱基分布图
覆盖均一性被认为是全面衡量测序数据质量最好的方式之一。与理论泊松分布图极为接近单碱基深度分布图,可真实地反应该样品数据的均一性高。通常来说,数据量越大,冗余越多,随机性也会相应地降低,因此与理论泊松分布图的偏差会测序深度的增加而增加(表3)。30X的数据,其泊松分布图SD值在2.0左右都可说明其数据均一性高。

5 不同平均测序深度累积分布

注:4X是衡量检测变异的一个重要指标,在30X时,4X占的比例大99.21%,与覆盖度接近。
6 不同平均覆盖深度与基因组覆盖率之间的关系
基因组覆盖率与平均碱基测序深度c之间的关系:
碱基的覆盖深度符合泊松分布,某个碱基没有被测序到的概率为P(X=0) = e-c ,这相当于整个基因组的未覆盖率,而基因组的覆盖率则为1- e-c
在保证shotgun打断完全随机的理想情况下,碱基的覆盖深度完全符合泊松分布图,在10X的时候,基因组覆盖率即可达到100%。在实际实验操作中,打断不可能完全随机,打断的随机性根据样品情况,打断片段大小而改变。打断片段越大,其随机性也越差。这就是为什么环化大片段文库的随机性要低于小片段文库。
基因组de novo组装中,根据基因组的GC含量、杂合度、重复序列及染色体倍数等因素把基因组分为简单基因组和复杂基因组。第一个被组装出来的人基因组在众多基因组中为较为简单且目前研究最为透彻的基因组之一,尽管如此,人的基因组中并不是绝对的完整染色体,仍然存在很多Gap和N区域,究其原因在于人的基因组中仍然存在很多高GC和低CG区域高度重复序列,如着丝粒区域等。由于这些区域的存在,全基因组重测序的随机性会与理论会有一定的偏差,且在测序过程中某些序列测不出来,因而,基因组覆盖度只能接近却达不到100%。
统计人全基因组重测序在不同的平均测序深度后得知,在5X, 15X,30X的基因组覆盖率平均值分别为96.31%, 99.56%, 99.72%(图4),可得知平均覆盖深度与基因组覆盖率成正比例相关,基因组覆盖率随测序深度的增加而增加,在15X之后趋于饱和。对于相同的样本,不论是肿瘤或正常样本,在30X测序深度时,其基因组覆盖度已接近于饱和(图5)。从30X到80X,数据量翻了近3倍,而其基因组覆盖率仅增加0.1%-0.2%。


7 不同染色体在不同平均覆盖深度下的分布情况
人染色体中,有些区域会因为GC含量偏高或含有高度重复序列,测不出来(读取为N碱基)或测序错误率高,导致所测的reads在与参考基因组比对不上,造成基因组覆盖率偏低等现象。如对于有些基因组重复序列多、GC偏低或偏高的物种,10X数据基因组覆盖率不到80%。
在人的不同染色体,高GC区域及高度重复序列的是同样也是不一样的,因此,每条染色体的实际覆盖度并不完全一致。
有些染色体中拥有更多与其他染色体上面片段同源性较高的序列。 比对策略中例如soap会将比对到多出的序列随机放在某个位置 ,如果这条染色体上拥有更对的片段有这样的特征就会有更大的几率将多重比对的序列定位到这个位置从而某些染色体出现深度偏高的情况。


不同平均覆盖深度下在1-22号染色体及XY的基因组覆盖率(A)与实际覆盖深度(B)分布图。该数据统计了30X(34个男性样本,58个30X女性样本),15X(42个15X男性和48女性样本),5X(67个男性和17个女性样本)等不同平均覆盖深度下在1-22号染色体及XY的基因组覆盖率。
从A图中可以看出,大于15X的时候在各染色体表现出高基因组覆盖率和均一性,而仅5X平均覆盖深度时,覆盖率明显下降,且在各染色体间覆盖率不均一,尤其是Y染色体的覆盖率较差。从覆盖率来看,全基因组测序建议至少15X的平均覆盖深度以减少序列的丢失。
从B图可以看出,在数据量很少的时候,在每一条染色体的平均覆盖深度并没有很明显的规律,而超过15X以后,其Chr10,16号等染色体平均覆盖深度增加,而Chr17,19等染色体平均覆盖深度减少。出现这种差异是由于不同染色体的特性所引起的,其均一性会受不同染色体的重复序列及CG含量影响。
8 不同平均覆盖深度下数据的比对
同一样本的map-rate和unique-rate随数据变化的差异不大。受肿瘤基因组变异较大的影响,其unique-rate相比较与normal样本偏低

9 不同平均覆盖深度下检测变异
人全基因组重测序的目的就是为了检测在不同个体间的变异情况,寻找变异与疾病及表型之间的关系。因而,变异的精确度和灵敏性成了检测变异的两个重要的指标。由于测序存在着不可避免的系统错误,每一个位点只有在足够的reads支持下才能确保正确性。
在SNP calling中,检测出一个SNP位点最起码需要4条reads。4X以上的平均测序深度是数据量是否足够的一项重要指标。
为什么标准产品平均覆盖深度推荐30X?
1. 在30X的时候,大于4X的比例达99.21%,并趋于饱和
2. 杂合SNP的数目趋于饱和。在08年的时候,Nature的一篇文献上就对测序深度与变异间的关系做了相关的数据评估。研究发现,在15X的时候,纯和SNP趋于饱和。在30X的时候,杂合SNP趋于饱和(图7)
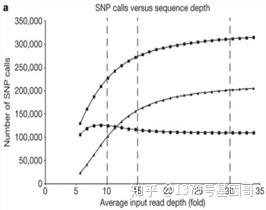
步证实30X测序深度对人全基因组重测序检测变异是否有很好的支持,8X,30X,80X3个梯度,分别进行评估:

从图8中可以看出,随深度的增加,其SNP杂合率逐渐减低,在30X之后,变化再显著。杂合SNP在较低深度下calling时,有很多位点仅有几条reads数支持,易受测序错误影响,对测序错误碱基和杂合变异碱基区分度低,从而将纯合SNP误读成杂合SNP。30X到80X,SNP的杂合率大多不再变化,也说侧面说明,SNP calling在30X时受测序错误影响小。
以80X为黄金标准(即假定在80X时,SNP calling都是正确的),分别用两种软件GATK和SAMtools对8X和30X的SNP和INDEL检测进行评估。
SN (sensitivity) and SP (specificity) 定义如下:
SN = n TP / (n TP +n FN )
SP = n TP / (n TP +n FP )
n TP : true positive个数, n FN : false negative个数, n FP : false positive个数.


从图9和图10可以看出,SNP calling在8X时的正确率约96%,在30X时的正确率为98%,仅增加2个百分点。而灵敏性则从8X时的80%增加到30X的98%。从中我们可以得知,SNP calling在低深度时,也可保持高精确度,随深度增加30X,精确度变化不大,灵敏性增加明显。SNP在30X时候,精确度和灵敏度均可达98%以上,随测序深度增加,可发现更多新的和稀有的突变。
Indel变异受不同软件的因素较大,但从8X到30X,精确度变化在1%左右,灵敏度同样呈现出大的上升趋势。因此也可以得出,随深度增加,精确度呈现细微变化,灵敏度增加明显。
10 总结
1. 单碱基分布图与泊松分布图的偏差随平均覆盖深度增加而增大。
2. 基因组覆盖率随随平均覆盖深度增加,在15X后增加不明显,30X时接近饱和。
3. Map-rate随平均覆盖深度变化,变化不显著。
4. 变异检测的专一性随覆盖深度增加而增加,在8X的时候,仍保持高专一性。
5. 变异检测的灵敏性随覆盖深度增加而增加明显。

