连锁不平衡分数回归 LD score regression -LDSC
本文包含以下三个部分:
- 理论基础
- 下载安装
- 使用教程
LDSC相关链接:
连锁不平衡 linkage disequilibrium LD
通过Bivariate LD Score regression估计遗传相关性 genetic correlation
基于功能分类分割遗传力 – 分层LD分数回归 Stratified LD score regression
1. 理论基础
什么是LD score?
SNP j的LD score可以被定义为该SNP与一定范围内其他SNP的r2之和。 LD score 衡量了该SNP标记的遗传变异性的大小。
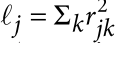
为什么要做 LD score regression?
在GWAS研究中,多基因性(polygenicity,即若干较小的基因效应)和干扰因素引起的偏差(如隐性关联 cryptic relatedness,群体分层population stratification等)都会造成检验的统计量的分布偏高(inflated)。但我们并不能分辨偏高的统计量到底是来自多基因性还是干扰因素,所以通过LD score regression,我们可以通过研究检验统计量与连锁不平衡(linkage disequilibrium)之间的关系来定量分析每部分的影响。
LDscore的原理?
GWAS检验中,对一个SNP效应量的估计通常也会包含与该SNP成LD的其他SNP的效应,也就是说一个与其他SNP成高LD的SNP,通常也会有更高的卡方检验量。
什么是LD score regression?
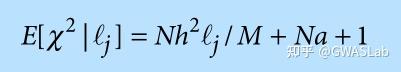
从LD score regression的结果中,我们可以得到怎样的信息?
如果我们用LD score对卡方统计量做回归,那么截距减1就是一个对干扰因素平均效应的估计值(estimator)。
2. 下载安装
接下来简单介绍如何进行LDscore regression。使用的软件为ldsc,可以从作者的github中拉取。ldsc为python脚本,clone了ldsc的库之后我们还需要利用anaconda配置环境,下载相关联的package。
ldsc: https://github.com/bulik/ldsc
git clone https://github.com/bulik/ldsc.git
cd ldsc
conda env create --file environment.yml
source activate ldsc以上步骤完成后,可以输入以下命令来检测是否安装成功
./ldsc.py -h./munge_sumstats.py -h除此以外,还需要相应群体的LD score,好消息是作者提供了处理好的欧洲 European 与 东亚 East asian的基于1000 genome的LD score以供我们使用,可以通过以下链接下载:
https://alkesgroup.broadinstitute.org/LDSCORE/
如果想使用其他的LD score则需要自行计算。j计算方法可参考:https://github.com/bulik/ldsc/wiki/LD-Score-Estimation-Tutorial
3. 使用教程
本教程改编自: https://github.com/bulik/ldsc/wiki/Heritability-and-Genetic-Correlation
主要介绍:1.the LD Score regression intercept for a schizophrenia GWAS 与 2.the SNP-heritability for schizophrenia
使用的数据来自 2013年发表的 PGC Cross-Disorder paper in the Lancet.,精神分裂症 sci 与 双相障碍 bip
3.1 准备步骤
下载LD score
wget https://data.broadinstitute.org/alkesgroup/LDSCORE/eur_w_ld_chr.tar.bz2
tar -jxvf eur_w_ld_chr.tar.bz2下载GWAS summary 统计量数据
wget www.med.unc.edu/pgc/files/resultfiles/pgc.cross.bip.zip
wget www.med.unc.edu/pgc/files/resultfiles/pgc.cross.scz.zip下载后解压,head命令查看文件是否正确
head pgc.cross.BIP11.2013-05.txt
snpid hg18chr bp a1 a2 or se pval info ngt CEUaf
rs3131972 1 742584 A G 1.092 0.0817 0.2819 0.694 0 0.16055
rs3131969 1 744045 A G 1.087 0.0781 0.2855 0.939 0 0.133028
rs3131967 1 744197 T C 1.093 0.0835 0.2859 0.869 0 .
rs1048488 1 750775 T C 0.9158 0.0817 0.2817 0.694 0 0.836449
rs12562034 1 758311 A G 0.9391 0.0807 0.4362 0.977 0 0.0925926
rs4040617 1 769185 A G 0.9205 0.0777 0.2864 0.98 0 0.87156
rs28576697 1 860508 T C 1.079 0.2305 0.7423 0.123 0 0.74537
rs1110052 1 863421 T G 1.088 0.2209 0.702 0.137 0 0.752294
rs7523549 1 869180 T C 1.823 0.8756 0.4929 0.13 0 0.0137615
head pgc.cross.SCZ17.2013-05.txt
snpid hg18chr bp a1 a2 or se pval info ngt CEUaf
rs3131972 1 742584 A G 1 0.0966 0.9991 0.702 0 0.16055
rs3131969 1 744045 A G 1 0.0925 0.9974 0.938 0 0.133028
rs3131967 1 744197 T C 1.001 0.0991 0.9928 0.866 0 .
rs1048488 1 750775 T C 0.9999 0.0966 0.9991 0.702 0 0.836449
rs12562034 1 758311 A G 1.025 0.0843 0.7716 0.988 0 0.0925926
rs4040617 1 769185 A G 0.9993 0.092 0.994 0.979 0 0.87156
rs4970383 1 828418 A C 1.096 0.1664 0.5806 0.439 0 0.201835
rs4475691 1 836671 T C 1.059 0.1181 0.6257 1.02 0 0.146789
rs1806509 1 843817 A C 0.9462 0.1539 0.7193 0.383 0 0.6009173.2 第一步 数据清理 (munge_sumstats.py)
原始数据并不是ldsc所需要的.sumstats 格式,所以我们需要先清理并提取需要的数据。利用ldsc附带的munge_sumstats.py可以将原始数据转换成.sumstats 格式。
The ldsc .sumstats format requires six pieces of information for each SNP:
- A unique identifier (e.g., the rs number)
- Allele 1 (effect allele)
- Allele 2 (non-effect allele)
- Sample size (which often varies from SNP to SNP)
- A P-value
- A signed summary statistic (beta, OR, log odds, Z-score, etc)
wget https://data.broadinstitute.org/alkesgroup/LDSCORE/w_hm3.snplist.bz2
bunzip2 w_hm3.snplist.bz2
munge_sumstats.py \
--sumstats pgc.cross.SCZ17.2013-05.txt \
--N 17115 \
--out scz \
--merge-alleles w_hm3.snplist
munge_sumstats.py \
--sumstats pgc.cross.BIP11.2013-05.txt \
--N 11810 \
--out bip \
--merge-alleles w_hm3.snplist注意:有时数据量较大时程序会卡死,无响应状态,可以尝试在选项中加上 --chunksize 500000 以解决此问题
输出的log文件
Metadata:
Mean chi^2 = 1.229
Lambda GC = 1.201
Max chi^2 = 32.4
11 Genome-wide significant SNPs (some may have been removed by filtering).
Conversion finished at Mon Apr 4 13:21:29 2016
Total time elapsed: 16.07s 3.3 第二步 LD score regression
利用清理好的数据,我们可以开始进行LD score regression,使用的是主程序ldsc.py
ldsc.py \
--h2 scz.sumstats.gz \
--ref-ld-chr eur_w_ld_chr/ \
--w-ld-chr eur_w_ld_chr/ \
--out scz_h2--h2 :计算遗传力,参数为上一步处理好的数据文件名
--ref-ld-chr :使用的按染色体号分类的LD score文件名,参数为LD score文件所在文件夹的路径。默认情况下ldsc会在文件名末尾添加染色体号,例如 --ref-ld-chr eur_w_ld_chr/ 的意思就是使用 eur_w_ld_chr/1.l2.ldscore ... eur_w_ld_chr/22.l2.ldscore 这些文件。如果你的染色体号在其他位置,也可以使用@来告诉LDSC,例如 --ref-ld-chr ld/chr@ 。当然也可以用 --ref-ld 来指定一个整体的LD score文件。
--w-ld-chr:指定ldsc回归权重所用的LDscore文件,理论上对于SNPj的LD分数,应当包含这个SNP与其他所有SNP的R2之和,但实际操作中,LD score回归对于计算权重的SNP的选择并不敏感,所以一般情况下我们可以使用与 --ref-ld 与相同的文件。.
--out : 输出文件的路径与前缀
ldsc输出的log文件结尾就是我们所要的结果,
Total Observed scale h2: 0.5907 (0.0484)
Lambda GC: 1.2038
Mean Chi^2: 1.2336
Intercept: 1.0014 (0.0113)
Ratio: 0.0059 (0.0482)结果解读:
Total Observed scale h2: 总的观测尺度的遗传力 (详见易感性尺度遗传力与观测尺度遗传力 Liability scale heritability & observed scale heritability)
Lambda GC 与 Mean chi^2: 用于评估群体分层与隐性关联的影响
Ratio: 定义为attenuation ratio = (LDSC intercept – 1) / (mean χ2 – 1), 衰减比:用来估计混淆因素与遗传效应的相对平衡
参考:
Bulik-Sullivan, B., Loh, PR., Finucane, H. et al. LD Score regression distinguishes confounding from polygenicity in genome-wide association studies. Nat Genet 47, 291–295 (2015). https://doi.org/10.1038/ng.3211

