【Graph Embedding】node2vec:算法原理,实现和应用
前面介绍过基于DFS邻域的DeepWalk和基于BFS邻域的LINE。
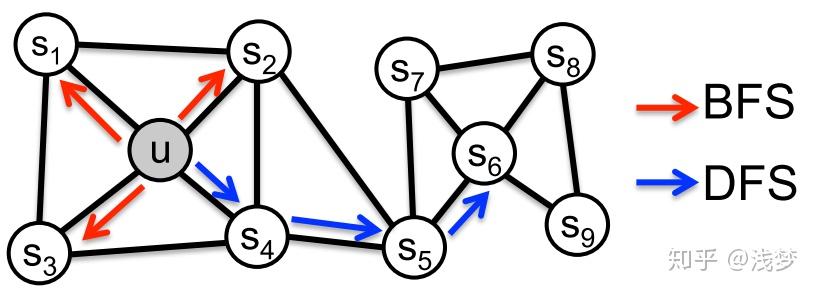
node2vec是一种综合考虑DFS邻域和BFS邻域的graph embedding方法。简单来说,可以看作是deepwalk的一种扩展,是结合了DFS和BFS随机游走的deepwalk。
nodo2vec 算法原理
优化目标
设 f(u) 是将顶点 u 映射为embedding向量的映射函数,对于图中每个顶点u,定义 N_S(u) 为通过采样策略 S 采样出的顶点 u 的近邻顶点集合。
node2vec优化的目标是给定每个顶点条件下,令其近邻顶点(如何定义近邻顶点很重要)出现的概率最大。
max_f {\sum_{u\in V}\log{Pr(N_S(U)|f(u))}}
为了将上述最优化问题可解,文章提出两个假设:
- 条件独立性假设
假设给定源顶点下,其近邻顶点出现的概率与近邻集合中其余顶点无关。 Pr(N_s(u)|f(u))=\prod_{n_i\in N_s(u)} Pr(n_i|f(u))
- 特征空间对称性假设
这里是说一个顶点作为源顶点和作为近邻顶点的时候共享同一套embedding向量。(对比LINE中的2阶相似度,一个顶点作为源点和近邻点的时候是拥有不同的embedding向量的) 在这个假设下,上述条件概率公式可表示为 Pr(n_i|f(u))=\frac{\exp{f(n_i)\cdot f(u)}}{\sum_{v\in V}{\exp{f(v)\cdot f(u)}}}
根据以上两个假设条件,最终的目标函数表示为 max_f{\sum_{u\in V}[-\log{Z_u}+\sum_{n_i\in N_s(u)}{f(n_i)\cdot f(u)}]}
由于归一化因子 Z_u=\sum_{n_i\in N_s(u)}{\exp(f(n_i)\cdot f(u))} 的计算代价高,所以采用负采样技术优化。
顶点序列采样策略
node2vec依然采用随机游走的方式获取顶点的近邻序列,不同的是node2vec采用的是一种有偏的随机游走。
给定当前顶点 v ,访问下一个顶点 x 的概率为
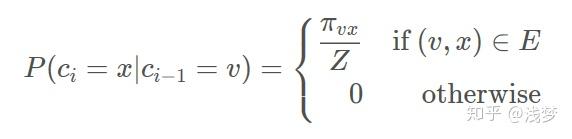
\pi_{vx} 是顶点 v 和顶点 x 之间的未归一化转移概率, Z 是归一化常数。
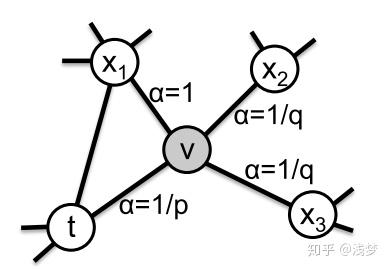
node2vec引入两个超参数 p 和 q 来控制随机游走的策略,假设当前随机游走经过边 (t,v) 到达顶点 v 设 \pi_{vx}=\alpha_{pq}(t,x)\cdot w_{vx} , w_{vx} 是顶点 v 和 x 之间的边权,
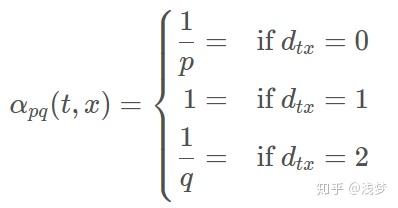
d_{tx} 为顶点 t 和顶点 x 之间的最短路径距离。
下面讨论超参数 p 和 q 对游走策略的影响
- Return parameter,p
参数p控制重复访问刚刚访问过的顶点的概率。 注意到p仅作用于 d_{tx}=0 的情况,而 d_{tx}=0 表示顶点 x 就是访问当前顶点 v 之前刚刚访问过的顶点。 那么若 p 较高,则访问刚刚访问过的顶点的概率会变低,反之变高。
- In-out papameter,q
q 控制着游走是向外还是向内,若 q>1 ,随机游走倾向于访问和 t 接近的顶点(偏向BFS)。若 q<1 ,倾向于访问远离 t 的顶点(偏向DFS)。
下面的图描述的是当从 t 访问到 v 时,决定下一个访问顶点时每个顶点对应的 \alpha 。
学习算法
采样完顶点序列后,剩下的步骤就和deepwalk一样了,用word2vec去学习顶点的embedding向量。 值得注意的是node2vecWalk中不再是随机抽取邻接点,而是按概率抽取,node2vec采用了Alias算法进行顶点采样。
node2vec核心代码

node2vecWalk
通过上面的伪代码可以看到,node2vec和deepwalk非常类似,主要区别在于顶点序列的采样策略不同,所以这里我们主要关注node2vecWalk的实现。
由于采样时需要考虑前面2步访问过的顶点,所以当访问序列中只有1个顶点时,直接使用当前顶点和邻居顶点之间的边权作为采样依据。 当序列多余2个顶点时,使用文章提到的有偏采样。
def node2vec_walk(self, walk_length, start_node):
G = self.G
alias_nodes = self.alias_nodes
alias_edges = self.alias_edges
walk = [start_node]
while len(walk) < walk_length:
cur = walk[-1]
cur_nbrs = list(G.neighbors(cur))
if len(cur_nbrs) > 0:
if len(walk) == 1:
walk.append(cur_nbrs[alias_sample(alias_nodes[cur][0], alias_nodes[cur][1])])
else:
prev = walk[-2]
edge = (prev, cur)
next_node = cur_nbrs[alias_sample(alias_edges[edge][0],alias_edges[edge][1])]
walk.append(next_node)
else:
break
return walk构造采样表
preprocess_transition_probs分别生成alias_nodes和alias_edges,alias_nodes存储着在每个顶点时决定下一次访问其邻接点时需要的alias表(不考虑当前顶点之前访问的顶点)。alias_edges存储着在前一个访问顶点为 t ,当前顶点为 v 时决定下一次访问哪个邻接点时需要的alias表。
get_alias_edge方法返回的是在上一次访问顶点 t ,当前访问顶点为 v 时到下一个顶点 x 的未归一化转移概率 \pi_{vx}=\alpha_{pq}(t,x)\cdot w_{vx}
def get_alias_edge(self, t, v):
G = self.G
p = self.p
q = self.q
unnormalized_probs = []
for x in G.neighbors(v):
weight = G[v][x].get('weight', 1.0)# w_vx
if x == t:# d_tx == 0
unnormalized_probs.append(weight/p)
elif G.has_edge(x, t):# d_tx == 1
unnormalized_probs.append(weight)
else:# d_tx == 2
unnormalized_probs.append(weight/q)
norm_const = sum(unnormalized_probs)
normalized_probs = [float(u_prob)/norm_const for u_prob in unnormalized_probs]
return create_alias_table(normalized_probs)
def preprocess_transition_probs(self):
G = self.G
alias_nodes = {}
for node in G.nodes():
unnormalized_probs = [G[node][nbr].get('weight', 1.0) for nbr in G.neighbors(node)]
norm_const = sum(unnormalized_probs)
normalized_probs = [float(u_prob)/norm_const for u_prob in unnormalized_probs]
alias_nodes[node] = create_alias_table(normalized_probs)
alias_edges = {}
for edge in G.edges():
alias_edges[edge] = self.get_alias_edge(edge[0], edge[1])
self.alias_nodes = alias_nodes
self.alias_edges = alias_edges
returnnode2vec应用
使用node2vec在wiki数据集上进行节点分类任务和可视化任务。 wiki数据集包含 2,405 个网页和17,981条网页之间的链接关系,以及每个网页的所属类别。 通过简单的超参搜索,这里使用p=0.25,q=4的设置。
本例中的训练,评测和可视化的完整代码在下面的git仓库中,
G = nx.read_edgelist('../data/wiki/Wiki_edgelist.txt',create_using=nx.DiGraph(),nodetype=None,data=[('weight',int)])
model = Node2Vec(G,walk_length=10,num_walks=80,p=0.25,q=4,workers=1)
model.train(window_size=5,iter=3)
embeddings = model.get_embeddings()
evaluate_embeddings(embeddings)
plot_embeddings(embeddings)分类任务
micro-F1: 0.6757 macro-F1: 0.5917
这个结果相比于DeepWalk和LINE是有提升的。
可视化
这个结果相比于DeepWalk和LINE可以看到不同类别的分布更加分散了。

最后补充一个node2vec在业界的应用介绍
参考资料
为了方便大家学习,我把一些相关的经典文章和代码实现进行了打包汇总并放在了github仓库里,感兴趣的同学可以看看~
欢迎加入学习交流小组,一个由热爱分享与交流的小伙伴们共建维护的小圈子,专注于前沿算法技术与实践经验的分享学习。



